【分子克隆 】SnapGene 详细使用教程

SnapGene使用教程










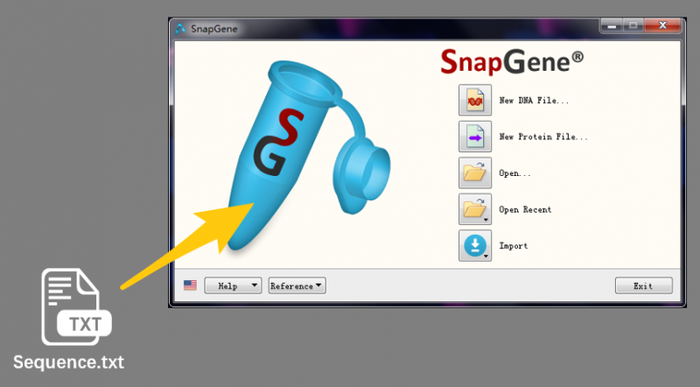
1 打开SnapGene,直接将txt拖入snapgene起始界面。
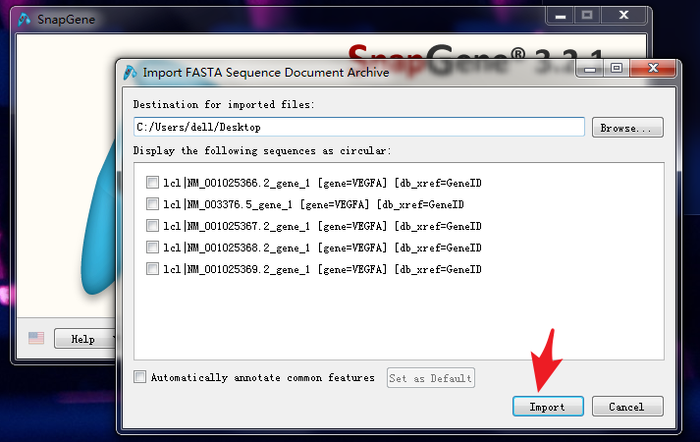
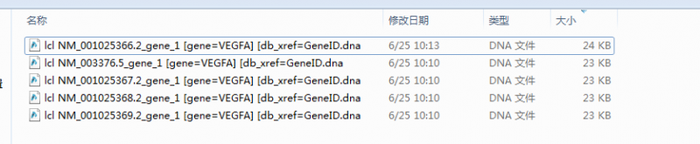
2 SnapGene将自动识别txt中的每个序列,并将其拆分成为单独的序列文件,点击“Import”,软件将生成一个文件夹,文件夹中含有txt中的每一个FASTA序列。
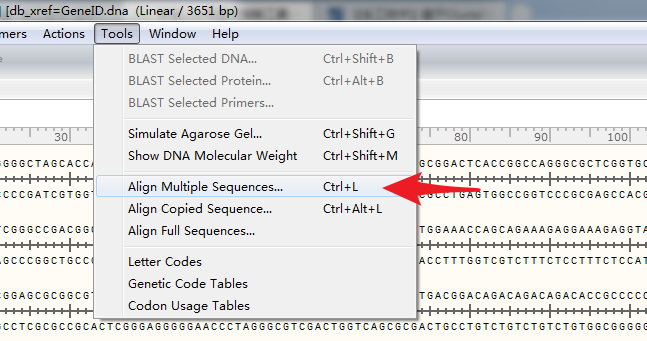
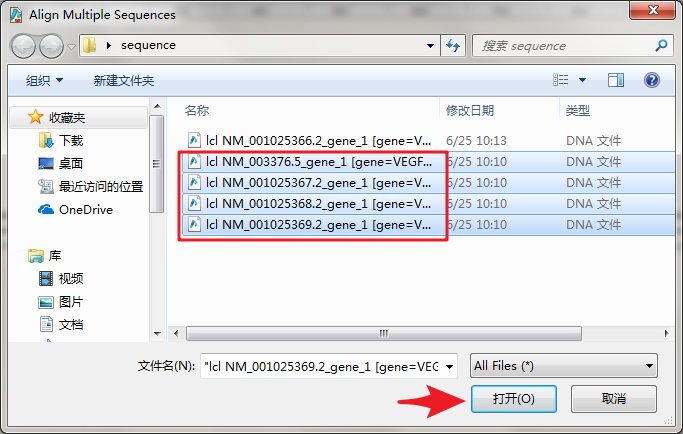
3. 用SnapGene打开任意一个序列(推荐打开文件大小最大的),选择Tools菜单栏中的“Align Multiple Sequences”功能(快捷键Ctrl+L)
4. 在弹出的窗口中,将剩下几个序列都选中,点击“打开”,SnapGene将对选中的序列进行多重比对:
5. 比对结果如下图所示,在下方的Map标签页,我们能够看到这几个转录变体同源及非同源区域所在的位置,非同源区域以空白或者三角显示,同源序列以蓝色显示。

6. 点击下方的Sequence标签页,我们能够看到具体的比对结果信息:
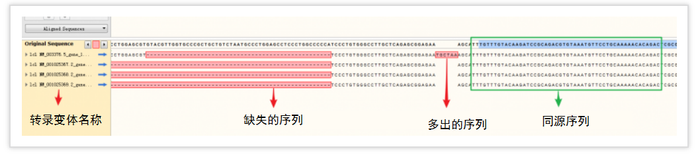
7. 我们可以用鼠标选中绿色的区域,复制、粘贴就得到了这几个转录变体的同源序列了。







 然后在该序列5’端上添加数个碱基作为保护碱基。点击完成。
然后在该序列5’端上添加数个碱基作为保护碱基。点击完成。


以pEGFP-C1构建human p53 CDS过表达载体为例,通过酶切位点筛选,选择“EcoRI”和“BamHI”两种内切酶,其中需要注意的是:EcoRI在上游,BamHI在下游
1.按照之前步骤,用snapgene打开p53 CDS序列
2.选择底部“Sequence”页面
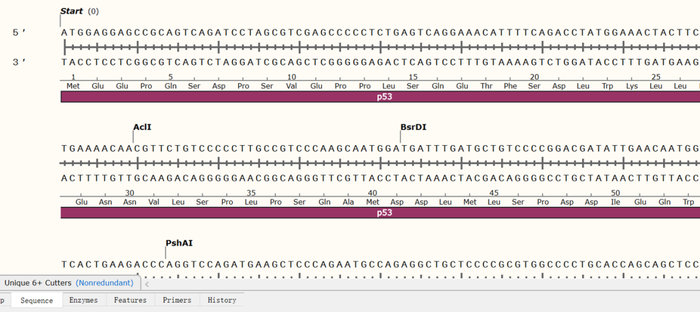
3.接下来进行引物设计:首先先选上游前20个碱基
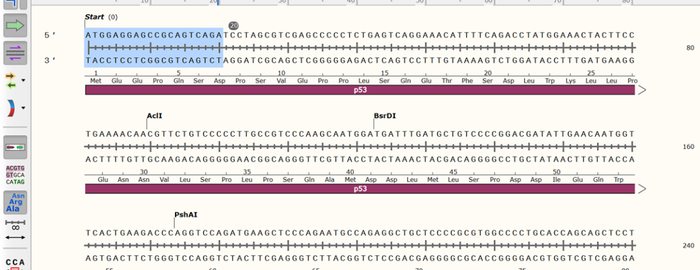
4.点击“Primers”→“add primer”,在弹出的选项框中选择“TOP strand”
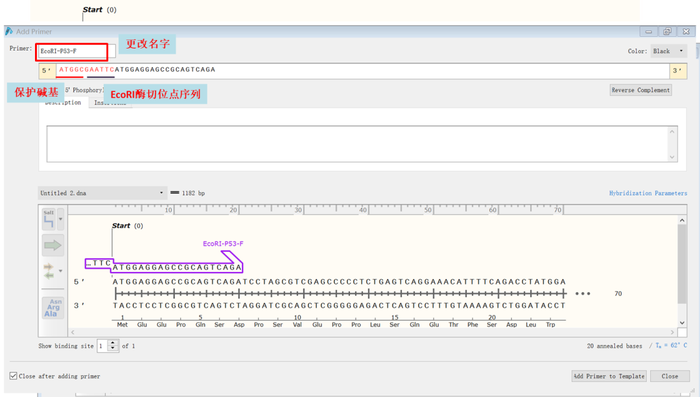
5.按下图所示,更改上游primer的名字以及添加酶切位点序列(百度或用snapgene软件打开载体序列均可查找到内切酶对应的切割序列)和保护碱基的序列,然后点击“Add primer to template”
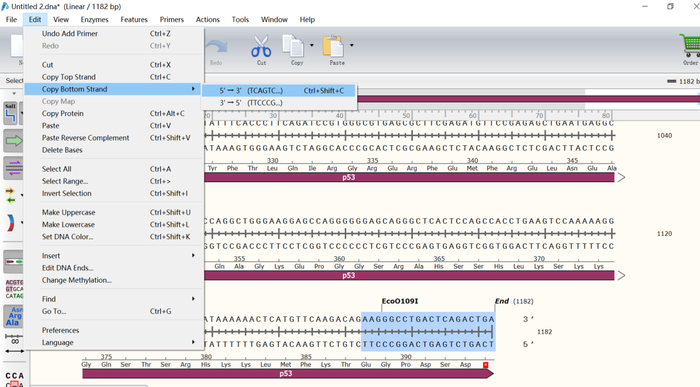
6.然后选择下游20个碱基,点击“Edit”→“copy bottom strand”,选择“5’→3’”
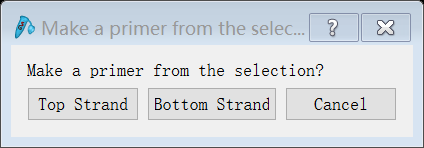
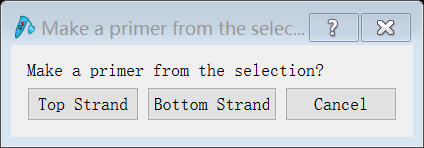
7.然后,点击“Primers”→“Add primer”,在弹出的选项框中点击右上角的×,直接关闭
8.将序列粘贴到序列框中,同理更改名字以及添加酶切位点序列(下游酶切位点是BamHI)和保护碱基的序列,然后点击“add primer to template”
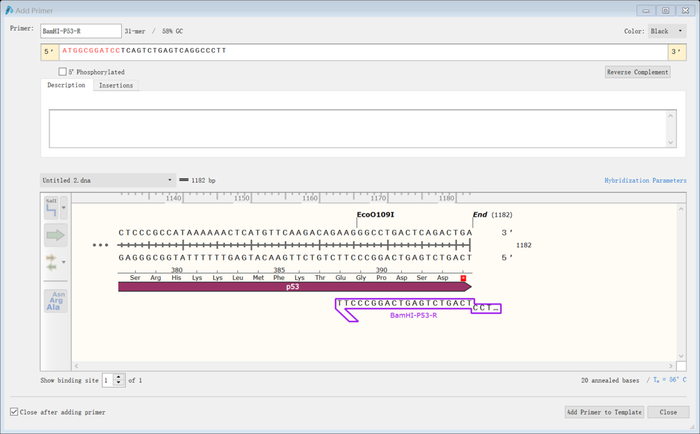
9.点击“Map”,Ctrl键选择两个引物(如图),点击“Action”→“PCR”
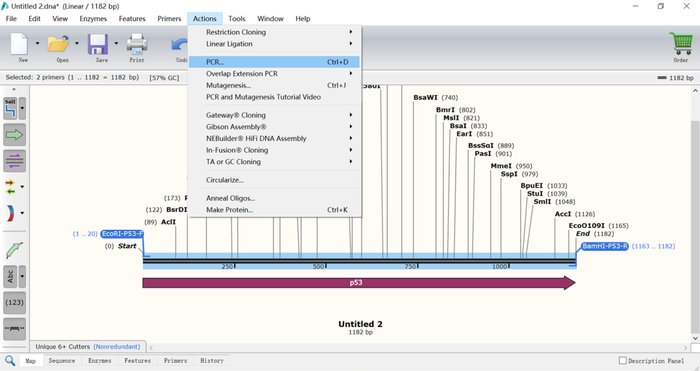
10.点击“PCR”,即获得扩增产物(如下图所示)
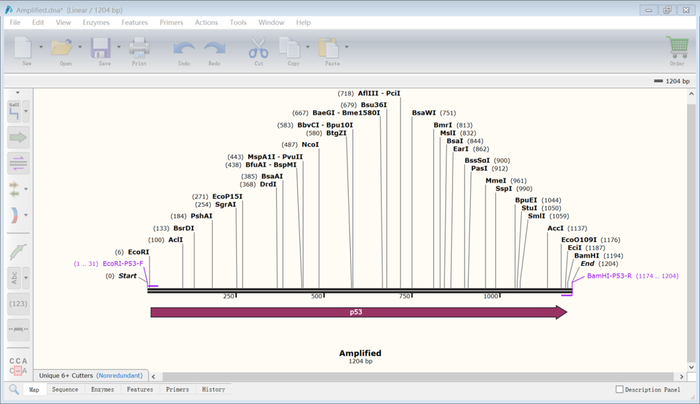
11.打开表达载体序列,按Ctrl键选择两个酶切位点(蓝色标记的),点击“Action”→“Restriction cloning”→“Insert fragment”
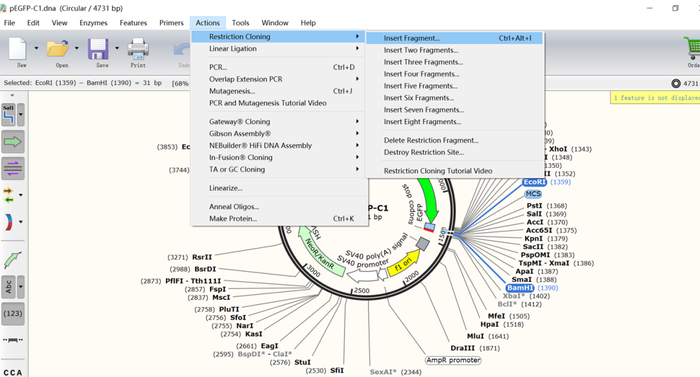
12.点击“Insert”,选择刚刚扩增产物的文件“Amplified.dna”,然后分别输入相应内切酶的名字,点击插入的片段
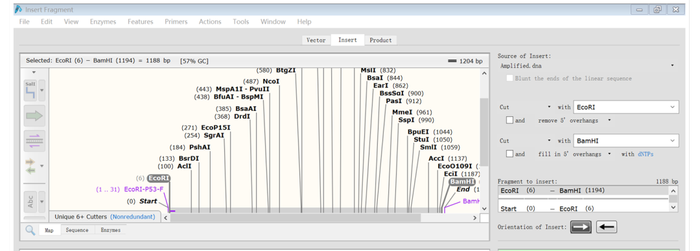
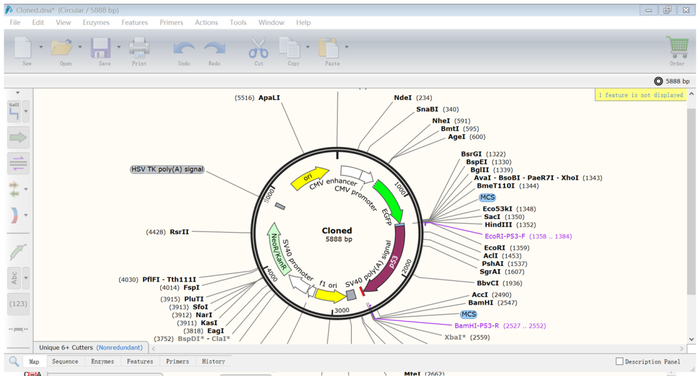
13.最后在右下角点击“Clone”即可,结果如下

科研鹿
展源
何发
相关文章
-

QC, IQC, IPQC, QA,到底是什么鬼?
2020-05-27
-

AAS法分析茶叶中的铅,镉,砷
2020-05-27
-
红外光谱分析,你了解多少?
2021-01-11
-

HPLC检测器,你了解吗?
2024-03-06
-

三聚氰胺,你还要害多少人
2020-05-27
-

超净工作台原理,使用与维护
2020-05-27
-

选对 ,快速开发方法
2020-05-27
-

'die','device','chip'有什么区别?
2024-02-21
-
检测有机氯类农药,气相色谱法检测法
2021-01-12

加载更多